このチュートリアルでは、Kaggleで公開されている「心不全予測」データセットについて学びます。GridDBを使用して、どのようにデータを抽出することができるかを見ていきます。その後、いくつかの探索的データ解析を行います。最後に、将来の予測を行うための機械学習モデルを構築します。このチュートリアルのアウトラインは以下の通りです。
- 環境のセットアップ
- データセットの紹介
- 必要なライブラリのインポート
- データセットの読み込み
- 探索的データ解析
- カテゴリ変数の取り扱い
- 機械学習モデル
- モデル評価
- 結論
- 参考文献
Jupyterファイルはこちらからご覧いただけます: https://github.com/griddbnet/Blogs/blob/main/Heart%20Failure%20Prediction.ipynb
1. 環境設定
このチュートリアルは、Jupyter Notebooks (Anaconda version 4.8.3) と Python version 3.8 on Windows 10 Operating system で実施されます。以下のパッケージは、コード実行の前にインストールする必要があります。
ハイパーリンクをクリックすると、インストールが開始されます。また、コマンドラインを使用している場合は、単に pip install package-name と入力してください。Anacondaの場合は、conda install package-nameと入力してもうまくいきます。
データセットをロードする際、このチュートリアルではGridDBを使用する方法とPandasを使用する方法の2つを取り上げます。Pythonを使用してGridDBにアクセスするには、以下のパッケージも事前にインストールしておく必要があります。
- GridDB C-クライアント
- SWIG (Simplified Wrapper and Interface Generator)
- GridDB Pythonクライアント
2. データセットの紹介
循環器疾患は、世界的な死因の1つです。そのため、機械学習で心不全予知ができれば、その貢献度は大きいと考えられます。このチュートリアルで使用するデータセットは、BMC Medical Informatics and Decision MakingのDavide Chicco, Giuseppe Jurmanによって開発されたものです。このデータセットはオープンソース化されており、Kaggleからダウンロードすることができます。
データは12の属性(または列)を持つ合計918のインスタンス(または行)を含んでいます。これらの12属性のうち、5つはカテゴリーで、7つは数値の性質を持っています。それでは、必要なライブラリをインポートしてみましょう。
3. 必要なライブラリのインポート
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.model_selection import train_test_split
import plotly.graph_objects as go
import plotly.exprs as px
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import classification_report
from sklearn.metrics import plot_confusion_matrixインストールに成功した場合、上記のセルはエラーメッセージや警告を出さずに正常に実行されるはずです。しかし、もしエラーが発生した場合は…
- インストールが成功したかどうか再確認してください。成功しなかった場合、
pip install package-nameを再度実行します。 - インストールされたパッケージのバージョンがあなたの anaconda/system のバージョンと互換性があるかどうかをチェックします。
4. データセットのロード
4.1 GridDBの利用
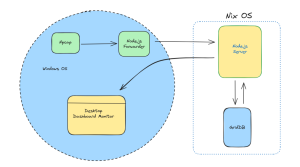
GridDBはスケーラブルでインメモリなNo SQLデータベースで、大量のデータを簡単に保存することができます。GridDBのpython-clientを使って、pandasのdataframeとしてpython環境に直接データを読み込むことができるようになりました。GridDBを初めて利用する場合は、GridDBへの読み書きのチュートリアルが参考になります。
データベースの設定が完了していることを前提に、データセットを読み込むためのSQLクエリをpythonで記述していきます。
import griddb_python as griddb
sql_statement = ('SELECT * FROM heart_failure_prediction')
heart_dataset = pd.read_sql_query(sql_statement, cont)変数 cont には、データが格納されるコンテナ情報が格納されます。
4.2 Pandasの使用
また、pandas の read_csv() 関数を使用することもできます。どちらの方法も、pandas のデータフレームの形式でデータをロードするため、同じ出力になることに注意してください。
heart_dataset = pd.read_csv('heart.csv')5. 探索的データ解析
まず、データセットの形状、すなわち行数と列数を決定しよう。
heart_dataset.shape(918, 12)
ここで、データがどのように見えるかの概要を知るために、pandasの head 関数を使用してデータの最初の5行を表示します。
heart_dataset.head()| Age | Sex | ChestPainType | RestingBP | Cholesterol | FastingBS | RestingECG | MaxHR | ExerciseAngina | Oldpeak | ST_Slope | HeartDisease | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 40 | M | ATA | 140 | 289 | 0 | Normal | 172 | N | 0.0 | Up | 0 |
| 1 | 49 | F | NAP | 160 | 180 | 0 | Normal | 156 | N | 1.0 | Flat | 1 |
| 2 | 37 | M | ATA | 130 | 283 | 0 | ST | 98 | N | 0.0 | Up | 0 |
| 3 | 48 | F | ASY | 138 | 214 | 0 | Normal | 108 | Y | 1.5 | Flat | 1 |
| 4 | 54 | M | NAP | 150 | 195 | 0 | Normal | 122 | N | 0.0 | Up | 0 |
可視化できました。このデータセットには、カテゴリ値と数値が混在しています。カテゴリ変数を直接機械学習モデルに渡すことはできないことに注意してください。モデル学習の前にエンコードする必要があります。続いて、属性のデータ型を確認しましょう。
heart_dataset.dtypesAge int64
Sex object
ChestPainType object
RestingBP int64
Cholesterol int64
FastingBS int64
RestingECG object
MaxHR int64
ExerciseAngina object
Oldpeak float64
ST_Slope object
HeartDisease int64
dtype: object
属性のうち5つはデータ型が「object」であり、カテゴリカルであることを表しています。残りの属性はfloatまたはintで、モデルの学習時に直接渡すことができます。
また、NULL値があると数学演算の際にエラーとなるため、NULL値は削除します。
heart_dataset.isna().sum()Age 0
Sex 0
ChestPainType 0
RestingBP 0
Cholesterol 0
FastingBS 0
RestingECG 0
MaxHR 0
ExerciseAngina 0
Oldpeak 0
ST_Slope 0
HeartDisease 0
dtype: int64
幸いなことに、NULL値はありません。機械学習のパートに移る前に、カテゴリ変数を探索します。
categorical_cols= heart_dataset.select_dtypes(include=['object'])
categorical_cols.columnsIndex(['Sex', 'ChestPainType', 'RestingECG', 'ExerciseAngina', 'ST_Slope'], dtype='object')
for cols in categorical_cols.columns:
print(cols,'-', len(categorical_cols[cols].unique()),'Labels')Sex - 2 Labels
ChestPainType - 4 Labels
RestingECG - 3 Labels
ExerciseAngina - 2 Labels
ST_Slope - 3 Labels
1つのCSVファイルなので、データセットをtrainとtestに分割しておくと、後々精度を計算するためにテストデータセットを残しておくことができます。ここでは、trainとtestの比率を70-30%としています。random_state` 変数は、インスタンスがランダムに選ばれ、偏りや歪みを最小化することを保証します。
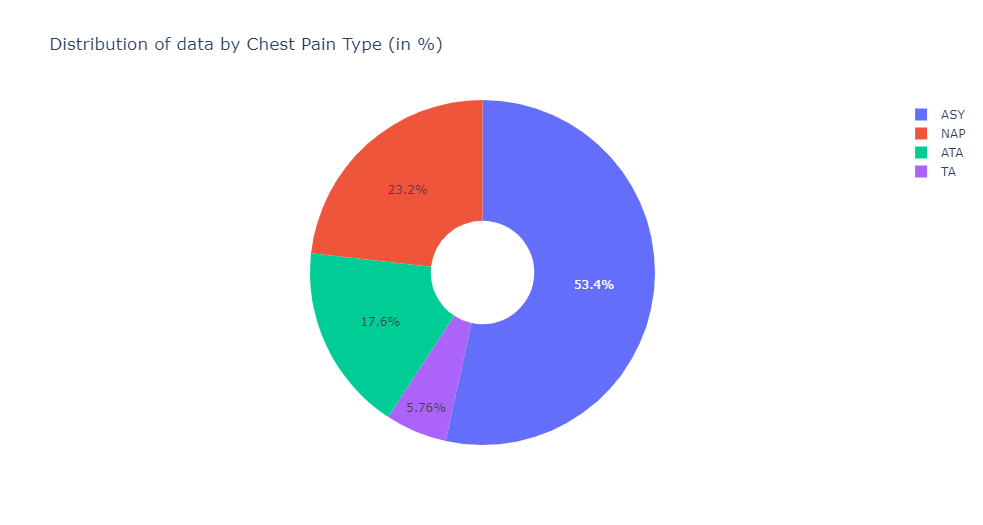
train, test = train_test_split(heart_dataset,test_size=0.3,random_state= 1234)labels = [x for x in train.ChestPainType.value_counts().index]
values = train.ChestPainType.value_counts()胸痛の種類別のデータ分布–。
fig = go.Figure(data=[go.Pie(labels=labels, values=values, hole=.3)])
fig.update_layout(
title_text="Distribution of data by Chest Pain Type (in %)")
fig.update_traces()
fig.show()さらに心臓病の有無に分けた性別ごとのデータ分布-。
fig=px.histogram(heart_dataset,
x="HeartDisease",
color="Sex",
hover_data=heart_dataset.columns,
title="Distribution of Heart Diseases by Gender",
barmode="group")
fig.show()
ヒストグラムやパイ関数を使って、他のカテゴリー変数で実験してみましょう。
6. カテゴリカル変数の取り扱い
5つのカテゴリカル属性のうち、SexとExerciseAnginaの2つの属性はバイナリ、つまり2つの値しか取らないことがわかりました。したがって、これらを 0 と 1 を使って手動でエンコードすることができます。他の値については、エンコード関数を使用します。
train['Sex'] = np.where(train['Sex'] == "M", 0, 1)
train['ExerciseAngina'] = np.where(train['ExerciseAngina'] == "N", 0, 1)
test['Sex'] = np.where(test['Sex'] == "M", 0, 1)
test['ExerciseAngina'] = np.where(test['ExerciseAngina'] == "N", 0, 1)<ipython-input-14-3d5da43d58db>:1: SettingWithCopyWarning:
A value is trying to be set on a copy of a slice from a DataFrame.
Try using .loc[row_indexer,col_indexer] = value instead
See the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy
<ipython-input-14-3d5da43d58db>:2: SettingWithCopyWarning:
A value is trying to be set on a copy of a slice from a DataFrame.
Try using .loc[row_indexer,col_indexer] = value instead
See the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy
<ipython-input-14-3d5da43d58db>:3: SettingWithCopyWarning:
A value is trying to be set on a copy of a slice from a DataFrame.
Try using .loc[row_indexer,col_indexer] = value instead
See the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy
<ipython-input-14-3d5da43d58db>:4: SettingWithCopyWarning:
A value is trying to be set on a copy of a slice from a DataFrame.
Try using .loc[row_indexer,col_indexer] = value instead
See the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy
train.head()| Age | Sex | ChestPainType | RestingBP | Cholesterol | FastingBS | RestingECG | MaxHR | ExerciseAngina | Oldpeak | ST_Slope | HeartDisease | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 578 | 57 | 0 | ASY | 156 | 173 | 0 | LVH | 119 | 1 | 3.0 | Down | 1 |
| 480 | 58 | 0 | ATA | 126 | 0 | 1 | Normal | 110 | 1 | 2.0 | Flat | 1 |
| 512 | 35 | 0 | NAP | 123 | 161 | 0 | ST | 153 | 0 | -0.1 | Up | 0 |
| 634 | 40 | 0 | TA | 140 | 199 | 0 | Normal | 178 | 1 | 1.4 | Up | 0 |
| 412 | 56 | 0 | ASY | 125 | 0 | 1 | Normal | 103 | 1 | 1.0 | Flat | 1 |
3つ以上の属性については、pandasの get_dummies 関数を使用します。これは、ラベルごとに新しい属性を作成します。例えば、ChestPainTypeは4つのラベルを持つので、0か1の値を持つ4つの新しい属性が作成されます。
train=pd.get_dummies(train)
test=pd.get_dummies(test)train.head()| Age | Sex | RestingBP | Cholesterol | FastingBS | MaxHR | ExerciseAngina | Oldpeak | HeartDisease | ChestPainType_ASY | ChestPainType_ATA | ChestPainType_NAP | ChestPainType_TA | RestingECG_LVH | RestingECG_Normal | RestingECG_ST | ST_Slope_Down | ST_Slope_Flat | ST_Slope_Up | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 578 | 57 | 0 | 156 | 173 | 0 | 119 | 1 | 3.0 | 1 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 |
| 480 | 58 | 0 | 126 | 0 | 1 | 110 | 1 | 2.0 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 |
| 512 | 35 | 0 | 123 | 161 | 0 | 153 | 0 | -0.1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 1 |
| 634 | 40 | 0 | 140 | 199 | 0 | 178 | 1 | 1.4 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 1 |
| 412 | 56 | 0 | 125 | 0 | 1 | 103 | 1 | 1.0 | 1 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 |
test.head()| Age | Sex | RestingBP | Cholesterol | FastingBS | MaxHR | ExerciseAngina | Oldpeak | HeartDisease | ChestPainType_ASY | ChestPainType_ATA | ChestPainType_NAP | ChestPainType_TA | RestingECG_LVH | RestingECG_Normal | RestingECG_ST | ST_Slope_Down | ST_Slope_Flat | ST_Slope_Up | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 581 | 48 | 0 | 140 | 208 | 0 | 159 | 1 | 1.5 | 1 | 1 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 |
| 623 | 60 | 0 | 140 | 293 | 0 | 170 | 0 | 1.2 | 1 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 |
| 60 | 49 | 0 | 100 | 253 | 0 | 174 | 0 | 0.0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 |
| 613 | 58 | 0 | 140 | 385 | 1 | 135 | 0 | 0.3 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 1 |
| 40 | 54 | 1 | 150 | 230 | 0 | 130 | 0 | 0.0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 |
train.shape(642, 19)
test.shape(276, 19)
エンコードした分、属性の総数が増えています。
ここで再び、トレーニングセットとテストセットを X と Y に分ける。Xは従属変数であるYの結果を決定する独立変数/属性の集合を表します。我々の場合、従属変数または説明変数はHeartDisease` になります。
x_train=train.drop(['HeartDisease'],1)
x_test=test.drop(['HeartDisease'],1)
y_train=train['HeartDisease']
y_test=test['HeartDisease']print(x_train.shape)
print(x_test.shape)(642, 18)
(276, 18)
7. 機械学習モデル
それでは、以下のパラメータでロジスティック回帰モデルを構築してみましょう。
- max_iter=10000`. ソルバーが収束するまでにかかる最大反復回数を示しています。デフォルトは100回です。
- penalty=l2
. ペナルティに使用するノルムを指定します。オプションは、None, l1, l2, and, elasticnet`です。デフォルトは l2 ですので、明示的に指定する必要はありません。
この関数では, class_weight, random_state, etc などのパラメータが利用可能です.使い方やデフォルトのパラメータを含む公式ドキュメントは こちらにあります。
lr = LogisticRegression(max_iter=10000)
model1=lr.fit(x_train, y_train)print("Train accuracy:",model1.score(x_train, y_train))Train accuracy: 0.8566978193146417
学習精度は約85.6%で、まずまずのスタートと言えそうです。それでは、テストデータセットに対して予測を行ってみましょう。
8. モデル評価
print("Test accuracy:",model1.score(x_test,y_test))Test accuracy: 0.894927536231884
テスト精度は89.5%と予想以上に高いです。素晴らしいですね。これでテストデータセットに predict メソッドを用いて予測値を格納できるようになりました。
lrpred = lr.predict(x_test)8.1 分類レポート
classification_report はモデル評価に使用される scikit-learn ライブラリの指標の一つです。この関数は以下を出力します。
Precision:True Positive/(True Positive+False Positive) として定義されます。Recall:True Positive/(True Positive+False Negative)で定義されます。F1 Score:精度と想起率の加重調和平均値。1は両者の加重が等しいことを意味します。Support:Ground Truth における各クラスの出現数。
print(classification_report(lrpred,y_test)) precision recall f1-score support
0 0.85 0.90 0.88 114
1 0.93 0.89 0.91 162
accuracy 0.89 276
macro avg 0.89 0.90 0.89 276
weighted avg 0.90 0.89 0.90 276
8.2 混同行列
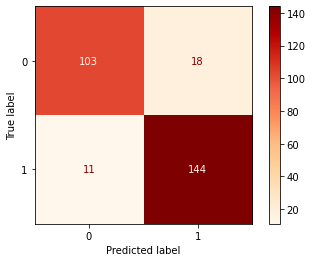
混同行列もまた、分類器を評価するために用いられる指標の1つです。定義によれば、混同行列の各エンティティ (i,j) は、実際にはグループ i に含まれるが、モデルによってグループ j に分類されるオブザベーションを表します。混同行列のパラメータをカスタマイズする方法について、詳しくは こちらを参照してください。
displr = plot_confusion_matrix(lr, x_test, y_test,cmap=plt.cm.OrRd , values_format='d')9. まとめ
このチュートリアルでは、GriDB と Python を使用して、心不全予測データセットの分類器を構築する方法を説明しました。データにアクセスする方法として、GridDBとPandasの2つを取り上げました。GridDBはスケーラブルでオープンソースであるため、大量のデータを扱う際に効率的な方法です。 ぜひGridDBをインストールしてください!
10. 参考文献
- https://www.kaggle.com/fedesoriano/heart-failure-prediction
- https://www.kaggle.com/sisharaneranjana/machine-learning-to-the-fore-to-save-lives
- https://www.kaggle.com/durgancegaur/a-guide-to-any-classification-problem
ブログの内容について疑問や質問がある場合は Q&A サイトである Stack Overflow に質問を投稿しましょう。 GridDB 開発者やエンジニアから速やかな回答が得られるようにするためにも "griddb" タグをつけることをお忘れなく。 https://stackoverflow.com/questions/ask?tags=griddb