はじめに
このドキュメントでは、まずGridDBの設定とRからの接続に関する注意点を説明します。 次に、The Cancer Genome Atlas (TCGA) から遺伝子発現データを取り込み、dplyrを使ってGridDBバックエンドに問い合わせ、簡単な要約統計を作成します。
前提条件
ここからソースコードをクローンすることでフォローすることが出来ます。 https://github.com/griddbnet/Blogs/tree/gene_analysis
git clone https://github.com/griddbnet/Blogs.git --branch gene_analysisGridDBのインストールと起動
GridDBをインストールするには、ドキュメントに記載されている手順に従うだけです。 Ubuntuマシンでは、debパッケージをダウンロードし、dpkg -iでインストールするのが最も簡単です。 その後、sudo systemctl start gridstoreでGridDBサービスを起動する必要があります。この後、データを読み込んでクエリを実行するために、Rからサービスに接続する準備が整いました。
Rの依存関係をインストールする
以下のコードを実行するには、いくつかのRパッケージのインストールが必要です(まだ利用可能でない場合)。以下では、このQuartoマークダウン文書で使用される依存関係を検出するために、attachmentパッケージを使っています。
attachment::att_from_rmd("01.Rmd")これらの依存関係は通常の install.packages メソッドでインストールすることが出来ます。ただし、Bioconductor パッケージには BiocManager::install が必要です。
deps <- attachment::att_from_rmd("01.Rmd")
install.packages(deps)または、Ubuntu環境では(クラウドセットアップではそうかもしれませんが)、Ubuntuのパッケージマネージャを使ってコンパイル済みのバージョンを入手するのがより早い方法かもしれません。
# replace `PKG1/2/3` with the required package names
sudo apt install r-cran-PKG1 r-cran-PKG2 r-cran-PKG3 ...RをローカルGridDBクラスタに接続する
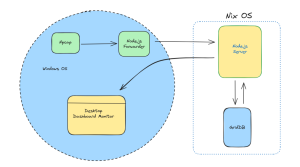
以前の記事で紹介しましたが、GridDBクラスタに接続するためにRJDBCパッケージ(Javaデータベース接続)を使用することにします。クラスタはローカルで動作しているので(インストールとスタート ガイドに従えばそうなります)、localhost の IP アドレス (127.0.0.1) とポート 20001 を必ず使用するようにしてください。
この2つのステップ、つまりjdbcドライバとGridDB接続の作成を、connect_to_griddbという関数にまとめると使いやすくなるはずです。RJDBCとDBIの名前空間全体を読み込んでいることに注意してください。探索的な対話セッションであれば問題ないかもしれませんが、より深刻なプロジェクトでは、もちろん R パッケージを作成して必要な関数だけをimportFrom`で明示的にインポートするか box などのアプローチを使って特定のインポートがあるモジュールを作成します。
suppressPackageStartupMessages({
library(RJDBC)
library(DBI)
library(dplyr)
library(tidyr)
library(purrr)
library(qs)
})
connect_to_griddb <- function() {
drv <- JDBC(driverClass = "com.toshiba.mwcloud.gs.sql.Driver",
# Point this to your gridstore jar
classPath = "~/src/jdbc/bin/gridstore-jdbc.jar")
conn <-
dbConnect(drv,
"jdbc:gs://127.0.0.1:20001/myCluster/public",
"admin",
"admin")
return(conn)
}データ
ここでは、The Cancer Genome Atlas Program (TCGA) で公開されているヒトのがん研究から得られたいくつかの遺伝子発現データを扱います。特に、BioconductorパッケージGSEABenchmarkeRによってパッケージ化された、遺伝子発現研究のベンチマークに使用することを目的としたデータに焦点を当てます。データはダウンロードされ、あらかじめ矩形集合にフォーマットされ、保存と読み込みを高速化するために qs アーカイブとして保存されています。
データセットをちょっと覗いてみましょう。
TCGA <- qs::qread("data/expressions.qs")
sapply(TCGA, nrow) |> summary()10個の癌の発現データがあり、平均1.2Mのオブザベーション(行)があります。各データセットには5つの列があり、
TCGA[[1]] |> head() |> knitr::kable()代表的ながんは以下の通りです。
sapply(TCGA, "[[", 1, 5) |> unlist() |> knitr::kable()GridDBにデータを読み込む
データを取り込むために、まず、上で定義した関数を使ってGridDBクラスタへの接続を作成します。次に、テーブルを作成する関数と、テーブルにデータを挿入する関数を定義します。 これらの関数は、データフレームのリストを循環させ、Rの*apply関数ファミリーやpurrrのmap_*関数ファミリーを用いて、各データフレームをシームレスにデータベースにロードすることができるようにします。
griddb <- connect_to_griddb()
create_table <- function(table_name) {
dbSendUpdate(
griddb,
sprintf(
"CREATE TABLE IF NOT EXISTS %s (Gene STRING, Sample STRING, Expression INTEGER, Code STRING, Name STRING);",
table_name
)
)
}
insert_table <- function(conn, name, df, append = TRUE) {
for (i in seq_len(nrow(df))) {
dbWriteTable(conn, name, df[i, ], append = append)
}
}
# using `invisible` here to hide non-informative output
invisible(lapply(names(TCGA), create_table))
DBI::dbListTables(griddb)
# 6 minutes for 10K rows per table. So 6 min for 100K rows
# 1h for 100K rows per table. So 1h for 1M rows.
invisible(pbapply::pblapply(seq_along(TCGA), function(i)
insert_table(
conn = griddb,
name = names(TCGA)[[i]],
df = TCGA[[i]][1:100000, ],
append = TRUE
))
)
DBI::dbDisconnect(griddb)griddb <- connect_to_griddb()
iter <- setNames(names(TCGA), names(TCGA))
map_dfr(iter, function(x) {
griddb |> tbl(x) |> collect() |> nrow()
}) |> knitr::kable()データ分析
簡単な統計データをまとめたものです。この時点では生物学的に意味のあるものはありません。主なアイデアは、dplyr、tidyr、purrrなどのおなじみのRパッケージを使って、高速なGridDBバックエンドデータベースに問い合わせることです。 以下のRコードは、MariaDBやPostgresSQLのデータベース用に開発されたものですが、GridDBのセットアップでも同様に動作することが出来ます。
平均発現量が2000を超えるがんデータセットごとの遺伝子
high_expression_genes <- purrr::map_dfr(iter, .id = "Cancer",
function(x) {
griddb |>
tbl(x) |>
group_by(Gene) |>
summarise(Mean_expression = mean(Expression, na.rm = TRUE)) |>
filter(Mean_expression >= 2000) |>
collect()
}
)
dim(high_expression_genes)
head(high_expression_genes) |> knitr::kable()平均発現量が300未満であるがんデータセットごとの遺伝子
low_expression_genes <- purrr::map_dfr(iter, .id = "Cancer",
function(x) {
griddb |>
tbl(x) |>
group_by(Gene) |>
summarise(Mean_expression = mean(Expression, na.rm = TRUE)) |>
filter(Mean_expression <= 300) |>
collect()
}
)
dim(low_expression_genes)
tail(low_expression_genes) |> knitr::kable()すべてのがんデータセットに共通する遺伝子
unique_genes_per_dataset <- purrr::map(iter,
function(x) {
griddb |>
tbl(x) |>
collect() |>
pull(Gene) |>
unique()
}
)
shared_genes <- Reduce(intersect, unique_genes_per_dataset)
shared_genes全データセットに共通する最初の10遺伝子の、がんデータセットごとの発現量の中央値
first10 <- shared_genes[1:10]
iter2 <- expand.grid(iter, first10, stringsAsFactors = FALSE)
first10_expression <-
purrr::map2_dfr(iter2$Var1, iter2$Var2, function(.x, .y) {
griddb |>
tbl(.x) |>
filter(Gene == .y) |>
collect()
})
first10_expression |>
group_by(Code, Name, Gene) |>
summarise(Mean_expression = mean(Expression, na.rm = TRUE)) |>
ungroup() |>
select(-Name) |>
pivot_wider(names_from = Code, values_from = Mean_expression) |>
knitr::kable()クリーンアップ
DBI::dbDisconnect(griddb)結論
このビネットでは、RからGridDBデータベースを利用する方法を紹介しました。10個のテーブルにある100万行の遺伝子発現データを約1時間で取り込み、DBIやdplyrといった使い慣れたツールを使ってシームレスにクエリを実行しました。
付録
遺伝子発現データのダウンロードと前処理に使用したR / Bioconductor コード。
# This has a lot of dependencies and can take a few minutes to install
# BiocManager::install("GSEABenchmarkeR")
library(GSEABenchmarkeR)
tcga <- loadEData("tcga", nr.datasets = 10)
cancer_abbreviations <- names(tcga)
cancer_names <- c("BLCA" = "Bladder Urothelial Carcinoma",
"BRCA" = "Breast Invasive Carcinoma",
"COAD" = "Colon Adenocarcinoma",
"HNSC" = "Head and Neck Squamous Cell Carcinoma",
"KICH" = "Kidney Chromophobe",
"KIRC" = "Kidney Renal Clear Cell Carcinoma",
"KIRP" = "Kidney Renal Papillary Cell Carcinoma",
"LIHC" = "Liver Hepatocellular Carcinoma",
"LUAD" = "Lung Adenocarcinoma",
"LUSC" = "Lung Squamous Cell Carcinoma")
expressions <- lapply(seq_along(tcga), function(i) {
tcga[[i]]@assays@data@listData |>
as.data.frame() |>
tibble::rownames_to_column(var = "Gene") |>
tidyr::pivot_longer(cols = -Gene, names_to = "Sample", values_to = "Expression") |>
mutate(cancer = cancer_abbreviations[[i]], cancer_name = cancer_names[[i]])
})
qs::qsave(expressions, "expressions.qs")セッション情報
sessionInfo()ブログの内容について疑問や質問がある場合は Q&A サイトである Stack Overflow に質問を投稿しましょう。 GridDB 開発者やエンジニアから速やかな回答が得られるようにするためにも "griddb" タグをつけることをお忘れなく。 https://stackoverflow.com/questions/ask?tags=griddb